Figure 4.
Télécharger l'image originale
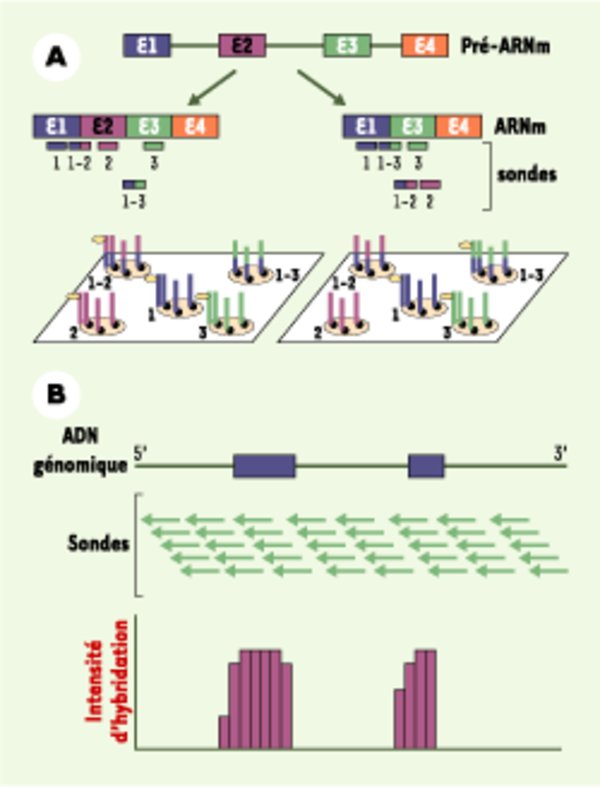
Identification des événements d’épissage alternatif. A. Utilisation de sondes oligonucléotidiques fixées sur des filtres à forte densité. Dans cet exemple, le pré-ARNm contient 4 exons, dont un exon alternatif (E2 en violet). Cinq types de sondes sont utilisées : 1, 2, 3, 1-2, 1-3 (par exemple, la sonde 1 s’hybride au niveau de l’exon 1, la sonde 1-2 s’hybride au niveau de la jonction entre exon 1 et exon 2…). Les sondes oligonucléotidiques sont fixées sur des filtres par leur région 3’ (symbolisée par un rond noir). Les ARN ou les produits de RT-PCR marqués sont ensuite hybridés sur ces filtres. L’ARN qui contient les exons 1, 2 et 3 s’est hybridé avec les sondes 1, 2, 3, et 1-2, tandis que l’ARN ne contenant pas l’exon 2 s’est hybridé avec les sondes 1, 3, et 1-3. L’hybridation est représentée par le petit ovale jaune. B. Hybridation moléculaire. À partir d’ADN génomique, une série de sondes (flèches vertes) est produite. Elles sont ensuite hybridées avec des populations d’ARNm ou d’ADN complémentaire. L’hybridation n’a lieu qu’avec les séquences exoniques (adapté de [15]).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




