Figure 1.
Télécharger l'image originale
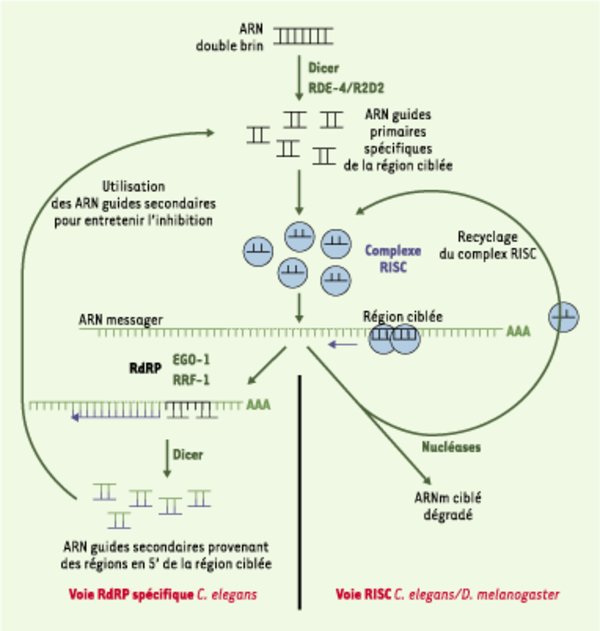
Mécanisme hypothétique de l’interférence par l’ARN chez C. elegans. Après son incorporation dans l’animal, l’ARN double brin est découpé en molécules d’ARN de 21 nucléotides [45]. Ces molécules sont considérées comme l’agent interférant à proprement parler [25] et ont été appelées ARN guides (short interfering RNA (siRNA)). Elles sont incorporées au sein d’un complexe ribonucléoprotéique, appelé RISC (RNA-induced silencing complex). Cette incorporation permet la localisation du complexe RISC au niveau de l’ARNm cible qui va être dégradé par Tudor-SN, une nucléase présente au sein du complexe (voie RISC) [34]. D’autres études ont permis d’établir l’existence potentielle de la voie RdRP (RNA-dependant RNA polymerase), une seconde voie de dégradation de l’ARNm cible [16]. Après interaction des ARN guides avec l’ARNm cible, un second brin d’ARN complémentaire à l’ARN messager serait synthétisé. La molécule d’ARN double brin, ainsi formée, serait à son tour, clivée en ARN guides par l’enzyme Dicer, et les ARN guides néo-formés (ou secondaires, par opposition aux ARN guides primaires qui proviennent directement de la dégradation du signal) seraient utilisés pour entretenir l’inhibition. Les ARN polymérases dépendantes de l’ARN (EGO-1, RRF-1) nécessaires à la synthèse du brin d’ARN complémentaire ainsi que les ARN guides secondaires n’ont été mis en évidence que chez C. elegans, ce qui suggère que la voie RdRP est spécifique de cet organisme (d’après [16]).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




