Figure 1.
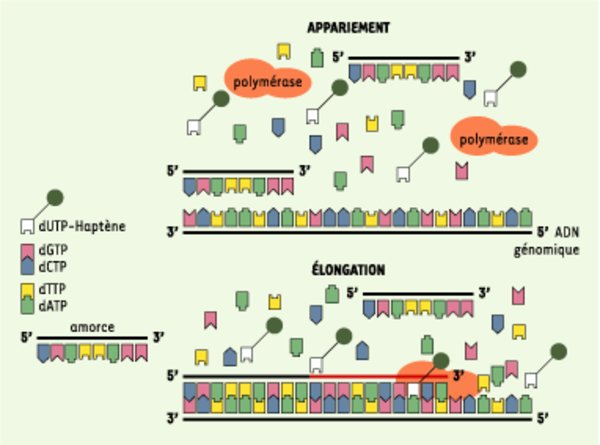
Schéma illustrant les deux étapes de la technique PRINS (marquage non isotopique par synthèse in situ amorcée) et ses principales applications. Un volume d’environ 45 μl d’un mélange réactionnel constitué entre autre d’une amorce (courte séquence oligonucléotidique de 18 à 35 nucléotides), de trois nucléotides non marqués et d’un nucléotide marqué (soit avec un haptène non fluorescent comme la biotine ou la digoxigénine, soit avec un fluorochrome) et de la Taq ADN polymérase est ajouté sur la lame. La première étape consiste en l’appariement des amorces non marquées à leur séquence d’ADN génomique complémentaire. La deuxième étape est une polymérisation à partir de l’extrémité 3’ du segment apparié par l’intermédiaire de la Taq ADN polymérase dont la fonction est de catalyser la réaction d’élongation. La présence du nucléotide marqué (dUTP sur la figure) permet la détection du fragment d’élongation nouvellement synthétisé. Dans le cas d’un couplage avec un haptène, la détection est faite à l’aide de différents systèmes de révélation (avidine-biotine, anticorps antidigoxigénine…). Dans le cas d’un marquage par un fluorochrome, la détection est directe. La durée de réalisation de la technique incluant la dénaturation des lames, la préparation des réactifs, les lavages et la révélation est d’environ 60 à 120 minutes. Les échantillons classiques d’exploitation du PRINS sont des préparations cytogénétiques, mais il est possible de réaliser ce type de réaction sur des coupes histologiques effectuées sous cryostat et sur des préparations de fibres d’ADN.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




