Figure 4.
Télécharger l'image originale
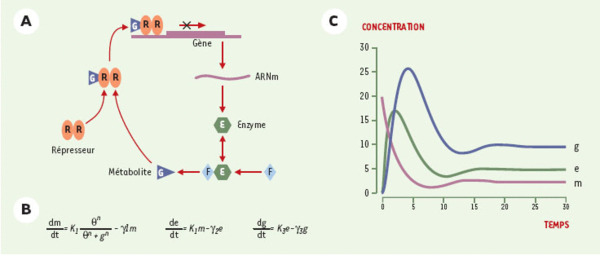
Modélisation différentielle du réseau correspondant au cas classique d’inhibition d’une réaction par son produit (d’après [26]). A. Schéma réactionnel. E (pour « enzyme ») et R (pour répresseur) représentent des protéines, et F et G représentent des métabolites. L’association du répresseur (sous forme de dimère) au métabolite G permet la fixation du complexe en amont du gène codant pour l’enzyme E et le blocage de la transcription de celui-ci. B. Système d’équations différentielles correspondant : m, e et g représentent les concentrations de l’ARNm codant pour l’enzyme, de la protéine enzymatique, et du métabolite G (corépresseur), respectivement. km, ke et kg sont des constantes de synthèses, alors que γm, γe et γg sont des constantes de dégradation, θ est une constante de seuil, et n une constante de coopérativité. C. Résultat d’une simulation pour un choix de conditions initiales et des valeurs paramétriques raisonnables mais arbitraires. Les trois courbes correspondent aux nombres de molécules d’ARNm (variable m, violet), de protéine enzymatique (variable e, vert), et du métabolite G (co-répres-seur, variable g, bleu). On obtient un état stationnaire stable.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




