Figure 1.
Télécharger l'image originale
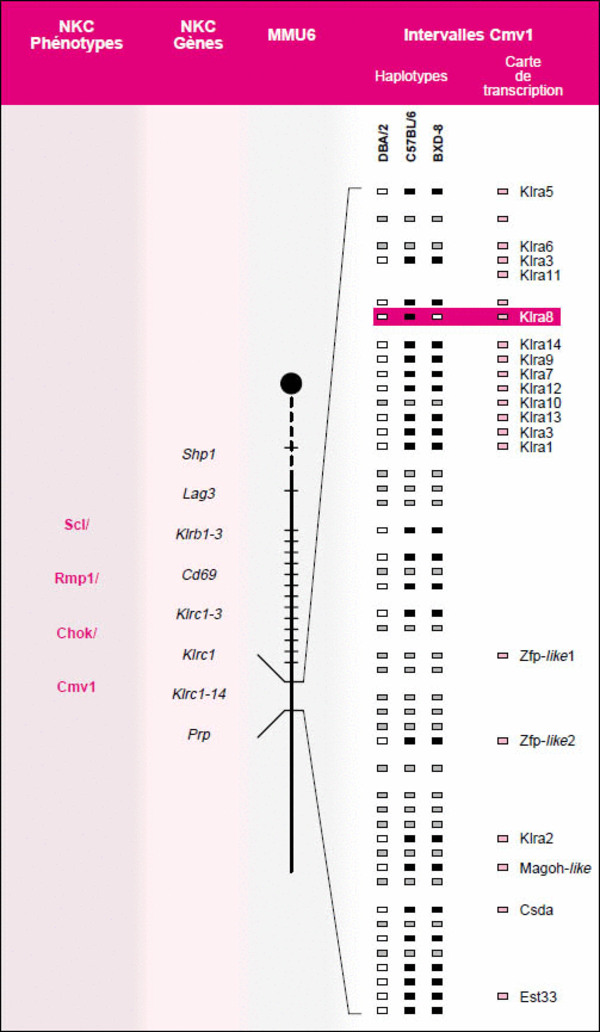
Cartographie du Complexe génique de la cellule NK (NKC) et du locus Cmv1.Le NKC est cartographié dans la région distale du chromosome 6 de la souris (MMU6). Cette région comprend des gènes codant pour des protéines importantes pour la reconnaissance du soi et l’élimination de cellules anormales comme les KLR (voir texte), la phophatase hématopoïétique Shp1 et le récepteur Lag3 (lymphocyte-activation gene 3), ainsi que des locus définis phénotypiquement à partir des fonctions de défense de l’hôte (phénotypes, en rouge). Chok contrôle l’activité tumoricide des cellules NK, tandis que Scl contrôle la susceptibilité au Leishmania major, Rmp1 au virus ectromelia et Cmv1 au cytomégalovirus. L’intervalle génétique pour Cmv1, défini par Klrd1 et Prp, est agrandi à droite de la figure. Cet intervalle correspond à 1,6 Mb d’ADN génomique. Les haplotypes pour les lignés sensibles (DBA/2 et BXD-8) et résistantes (C57BL/6) à l’infection ont été construits par génotypage des sondes poly-morphes (rectangles blancs, origine DBA/2 et noirs, origine C57BL/6) et non-polymorphes (rectangles gris). Les marqueurs sont cartographiés vis-à-vis d’une carte de transcription montrant les gènes candidats pour Cmv1 (rectangles roses). Le grand rectangle rouge indique la position du gène Klra8 dont l’absence détermine la susceptibilité au cytomégalovirus.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




