| Numéro |
Med Sci (Paris)
Volume 40, Numéro 6-7, Juin-Juillet 2024
|
|
|---|---|---|
| Page(s) | 498 - 500 | |
| Section | Nouvelles | |
| DOI | https://doi.org/10.1051/medsci/2024067 | |
| Publié en ligne | 8 juillet 2024 | |
Autoassemblage de la partie C-terminale transmembranaire de la protéine nsp3 du virus SARS-CoV-2
Self-assembly of the C-terminal and transmembrane region of the SARS-CoV-2 protein nsp3
Université Paris Saclay, CEA, CNRS UMR 9198, Institut de biologie intégrative de la cellule (I2BC), Gif-sur-Yvette, France
Les virus ne peuvent pas se répliquer de manière autonome. Une fois entrés dans la cellule hôte, ils doivent exploiter les ressources et les organites de la cellule pour copier leur matériel génétique et produire de nouvelles particules virales. Chaque type de virus élabore sa propre stratégie de réplication. Ainsi certains virus dont le génome est constitué d’ARN simple brin de polarité positive1 mobilisent et modifient les membranes de la cellule hôte pour élaborer des structures intracellulaires spécialisées qui servent d’usines de réplication pour le génome viral. Ces structures offrent ainsi un environnement optimal pour concentrer la machinerie de réplication et protéger le matériel génétique et les protéines virales contre les mécanismes de défense de la cellule hôte. Le remodelage membranaire est donc un aspect essentiel de leur stratégie de survie et de propagation dans l’organisme infecté. De nombreux virus pathogènes pour l’homme font partie de cette classe, comprenant entre autres les virus de la famille des Flaviviridae (dont le virus de l’hépatite C, le virus de la dengue ou le virus Zika), ou les virus de la famille des Coronaviridae (dont le SARS-CoV-2, responsable de la pandémie de COVID-19).
Dans le cas du SARS-CoV-2 et des coronavirus en général, le processus de remodelage membranaire conduit à la formation de compartiments membranaires spécifiques appelés « vésicules à double membrane ». Afin de pouvoir développer des thérapies antivirales ciblant le cycle de réplication des coronavirus, il est essentiel de comprendre les mécanismes moléculaires à l’origine de la formation de ces structures et d’élucider le rôle et l’organisation des protéines virales impliquées.
Protéines virales impliquées dans le remodelage membranaire induit par le SARS-CoV-2
Comme tous les coronavirus, le SARS-CoV-2 possède un grand génome pour un virus à ARN positif (environ 30 kilobases), génome dont les deux premiers tiers comportent deux cadres de lecture ouverts (open reading frames, ORF)qui conduisent à la synthèse de polyprotéines [1]. Deux activités protéase portées par le virus permettent de cliver ces polyprotéines en protéines non structurales individuelles (non structural proteins, nsp), qui sont essentielles à la réplication et au cycle de vie du virus. Le dernier tiers du génome code quatre protéines structurales, à savoir la protéine de surface Spike (S), la protéine de l’enveloppe (E), la protéine de l’enveloppe membranaire (M) et la protéine de la nucléocapside (N), ainsi que plusieurs protéines accessoires impliquées dans la modulation de la réponse de l’hôte à l’infection virale.
Plusieurs études ont mis en lumière l’implication de trois protéines non structurales des coronavirus (nsp3, nsp4 et nsp6) dans la biogénèse des vésicules à double membrane. Nous nous sommes intéressés à la protéine nsp3, qui est au cœur du processus de remodelage membranaire. Cette protéine possède une quinzaine de domaines fonctionnels, dont un domaine protéase (papain-like protease 2) qui permet son autoclivage ainsi que la libération de deux autres protéines non structurales, nsp1 et nsp2. Grâce à ses nombreux domaines et à sa capacité d’interagir avec d’autres protéines du virus et diverses protéines de la cellule hôte, nsp3 joue un rôle clé dans la réplication virale. En particulier, en interagissant avec nsp4, elle permet son recrutement aux sites de réplication virale, et cette étroite collaboration entre les deux protéines est essentielle au processus de remodelage membranaire. Un troisième partenaire, nsp6, est également requis pour une formation efficace des vésicules à double membrane [2]. Ces trois protéines sont les seules protéines non structurales du virus à présenter des segments transmembranaires, qui leur permettent de s’ancrer dans les membranes [3]. L’ARN viral synthétisé au sein de ces compartiments membranaires dédiés doit ensuite être exporté vers le cytoplasme de la cellule hôte pour être traduit en protéines virales ou incorporé dans de nouvelles particules virales. L’exportation de l’ARN se fait au travers de pores moléculaires récemment découverts dans les membranes des vésicules à double membrane. L’architecture de ces pores a d’abord été analysée par tomographie et cryomicroscopie électroniques dans des cellules infectées par un autre coronavirus, le virus de l’hépatite murine [4], puis, plus récemment, dans des cellules exprimant les deux protéines non-structurales nsp3 et nsp4 du SARS-CoV-2 [5]. Ces travaux ont permis d’analyser la structure tridimensionnelle des vésicules à double membrane avec une résolution suffisamment élevée pour démontrer la présence de pores moléculaires traversant la double membrane. Le composant majoritaire des pores serait la protéine nsp3. L’organisation de cette dernière et son autoassociation dans les pores seraient donc à l’origine de leur structure hexagonale ou hexamérique, et présentant une forme de couronne. Ainsi, six molécules de nsp3 seraient présentes par pore, et leurs extrémités N-terminales seraient orientées vers le cytoplasme, formant les pointes de la couronne, tandis que les parties C-terminales pointeraient vers l’intérieur du pore (Figure 1).
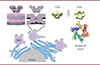 |
Figure 1. Organisation oligomérique de la partie C-terminale de la protéine nsp3 du virus SARS-CoV-2. Les vésicules à double membrane (double membrane vesicle, DMV), associées aux membranes du réticulum endoplasmique (RE) des cellules infectées par le coronavirus SARS-CoV-2, présentent des pores moléculaires en forme de couronne, qui traversent la double membrane et permettent l’exportation de l’ARN viral nouvellement synthétisé. La protéine virale nsp3 est le constituant majoritaire de ces pores. Une reconstruction 3D (avec une symétrie 6) a été réalisée à partir de l’analyse d’images de microscopie électronique d’un fragment C-terminal de la protéine nsp3 contenant les segments transmembranaires (nsp3C), qui a été synthétisé dans un système acellulaire. Deux perspectives sont présentées : une vue latérale et une vue de face de l’enveloppe de nsp3C reconstituée en nanodisques lipidiques (couleur jaune). L’enveloppe (vue latérale) est positionnée sur le pore moléculaire (représenté en violet, comme observé dans une coupe de cryotomographie électronique). Un modèle 3D de nsp3C obtenu à partir de la séquence d’acides aminés avec l’algorithme AlphaFold2 [6] (vue de face) est également présenté (avec une symétrie 6). |
Production et étude d’un fragment C-terminal transmembranaire de la protéine nsp3 du SARS-CoV-2
La protéine nsp3 apparaît donc comme une cible thérapeutique de choix pour développer des stratégies antivirales innovantes. Mais la taille et la complexité de cette longue protéine (1945 acides aminés) rendent son étude structurale très difficile. L’une des approches possibles consiste à considérer des portions structurées de la protéine (domaines) pour résoudre individuellement leur structure tridimensionnelle (3D) en utilisant les techniques de biologie structurale, telles que la cristallographie aux rayons X ou la résonance magnétique nucléaire (RMN). Les trois quarts de la protéine nsp3 ont ainsi été étudiés pour différents coronavirus, et les structures tridimensionnelles (3D) associées sont disponibles, mais les données structurales sur la partie C-terminale de nsp3, qui est celle qui contient les segments transmembranaires, sont encore très limitées. Un des objectifs de nos travaux de recherche est donc de comprendre comment la région C-terminale de nsp3 est structurée, et comment elle contribue à l’organisation des pores moléculaires qui traversent les vésicules à double membrane.
Au laboratoire, nous produisons les protéines dans un système d’expression acellulaire en utilisant des extraits de germe de blé. Ce système d’expression permet de produire in vitro des protéines généralement difficiles à produire dans des systèmes cellulaires classiques, comme les protéines membranaires. Grâce à un contrôle précis des conditions expérimentales, il permet également un criblage rapide des détergents afin de sélectionner ceux qui solubilisent le mieux les protéines membranaires2. Après sa production, sa solubilisation en présence d’un détergent, et sa purification, la région C-terminale de nsp3 (nsp3C) a ensuite été reconstituée dans des systèmes membranaires modèles avec des nanodisques lipidiques3 pour une analyse par microscopie électronique. Les images de coloration négative obtenues ont montré des objets présentant une organisation oligomérique en anneaux avec un pore central. La reconstruction tridimensionnelle montre un très bon accord avec la partie de nsp3 située dans la membrane extérieure (côté cytoplasme) des vésicules à double membrane (Figure 1). En parallèle, nous avons utilisé l’algorithme AlphaFold2, basé sur les méthodes d’« apprentissage profond » (deep learning), pour prédire la structure 3D de nsp3C à partir de sa séquence d’acides aminés [6], en imposant différents degrés d’autoassemblage, dont des hexamères pour prendre en compte la présence de six molécules de nsp3 par pore (Figure 1).
Perspectives
Le système de synthèse protéique in vitro utilisant des germes de blé s’est montré efficace pour produire la région C-terminale de nsp3 et obtenir des informations structurales sur cette partie de la protéine qui comprend les segments transmembranaires. En combinant les approches expérimentale et bio-computationnelle, nous avons montré que cette région est capable d’autoassemblage avec une organisation compatible avec une symétrie hexamérique [7]. Nous avons positionné la protéine dans les volumes 3D récemment obtenus pour les pores des vésicules à double membrane formés dans les cellules exprimant les deux protéines nsp3 et nsp4. Même si la protéine nsp3 apparaît comme étant le composant majoritaire des pores, son association avec nsp4 reste essentielle pour une formation efficace des vésicules et de leurs pores. Notre approche expérimentale étendue à l’étude du complexe nsp3-nsp4 devrait pouvoir nous éclairer sur l’organisation de ces deux protéines au sein des pores.
Liens d’intérêt
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Remerciements
Ce travail de recherche a bénéficié d’un financement de l’Université Paris-Saclay « programme de recherche exceptionnel UPSaclay suite à la crise sanitaire Covid-19 ». Les auteurs remercient l’ensemble des personnes qui ont contribué à ces travaux de recherche.
Les protéines membranaires sont souvent intégrées dans la bicouche lipidique des membranes biologiques, et sont alors très peu stables et souvent insolubles dans une solution aqueuse en raison de leur caractère hydrophobe. Afin de les maintenir en solution, l’une des approches consiste à utiliser des détergents. Le système acellulaire de synthèse protéique utilisant les extraits de germe de blé permet de cribler facilement différents détergents afin de déterminer celui qui permet de solubiliser le mieux la protéine membranaire considérée [8]
Les nanodisques lipidiques sont constitués d’une bicouche lipidique entourée par deux ceintures d’une protéine d’assemblage. Ils sont utilisés comme membranes artificielles pour préserver la structure et l’activité des protéines membranaires [9]
Références
- Lu R, Zhao X, Li J, et al. Genomic characterisation and epidemiology of novel coronavirus: implications for virus origins and receptor binding. Lancet 2019 ; 2020 : 395: 565–574. [Google Scholar]
- Ricciardi S, Guarino AM, Giaquinto L, et al. The role of NSP6 in the biogenesis of the SARS-CoV-2 replication organelle. Nature 2022; 606 : 761–78. [CrossRef] [PubMed] [Google Scholar]
- Oostra M, Hagemeijer MC, Van Gent M, et al. Topology and membrane anchoring of the coronavirus replication complex: not all hydrophobic domains of nsp3 and nsp6 are membrane spanning. J Virol 2008 ; 82 : 12392–12405. [CrossRef] [PubMed] [Google Scholar]
- Wolff G, Limpens R, Zevenhoven-Dobbe JC, et al. A molecular pore spans the double membrane of the coronavirus replication organelle. Science 2020; 369 : 1395–8. [CrossRef] [PubMed] [Google Scholar]
- Zimmermann L, Zhao X, Makroczyova J, et al. SARS-CoV-2 nsp3 and nsp4 are minimal constituents of a pore spanning replication organelle. Nat Commun 2023; 14 : 7894. [CrossRef] [PubMed] [Google Scholar]
- Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature 2021; 596 : 583–9. [CrossRef] [PubMed] [Google Scholar]
- Babot M, Boulard Y, Agouda S, et al. Oligomeric assembly of the C-terminal and transmembrane region of SARS-CoV-2 nsp3. J Virol 2024 ; 98 : e0157523. [CrossRef] [PubMed] [Google Scholar]
- Habersetzer J, Debbah M, Fogeron M-L, et al. In vitro translation of virally-encoded replication polyproteins to recapitulate polyprotein maturation processes. Protein Expr Purif 2020 ; 175 : 105694. [CrossRef] [PubMed] [Google Scholar]
- Denisov I-G, Sligar S-G. Nanodiscs for structural and functional studies of membrane proteins. Nat Struct Mol Biol 2016 ; 23 : 481–486. [CrossRef] [PubMed] [Google Scholar]
© 2024 médecine/sciences – Inserm
 Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Article publié sous les conditions définies par la licence Creative Commons Attribution License CC-BY (https://creativecommons.org/licenses/by/4.0), qui autorise sans restrictions l'utilisation, la diffusion, et la reproduction sur quelque support que ce soit, sous réserve de citation correcte de la publication originale.
Liste des figures
 |
Figure 1. Organisation oligomérique de la partie C-terminale de la protéine nsp3 du virus SARS-CoV-2. Les vésicules à double membrane (double membrane vesicle, DMV), associées aux membranes du réticulum endoplasmique (RE) des cellules infectées par le coronavirus SARS-CoV-2, présentent des pores moléculaires en forme de couronne, qui traversent la double membrane et permettent l’exportation de l’ARN viral nouvellement synthétisé. La protéine virale nsp3 est le constituant majoritaire de ces pores. Une reconstruction 3D (avec une symétrie 6) a été réalisée à partir de l’analyse d’images de microscopie électronique d’un fragment C-terminal de la protéine nsp3 contenant les segments transmembranaires (nsp3C), qui a été synthétisé dans un système acellulaire. Deux perspectives sont présentées : une vue latérale et une vue de face de l’enveloppe de nsp3C reconstituée en nanodisques lipidiques (couleur jaune). L’enveloppe (vue latérale) est positionnée sur le pore moléculaire (représenté en violet, comme observé dans une coupe de cryotomographie électronique). Un modèle 3D de nsp3C obtenu à partir de la séquence d’acides aminés avec l’algorithme AlphaFold2 [6] (vue de face) est également présenté (avec une symétrie 6). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




