Figure 2
Télécharger l'image originale
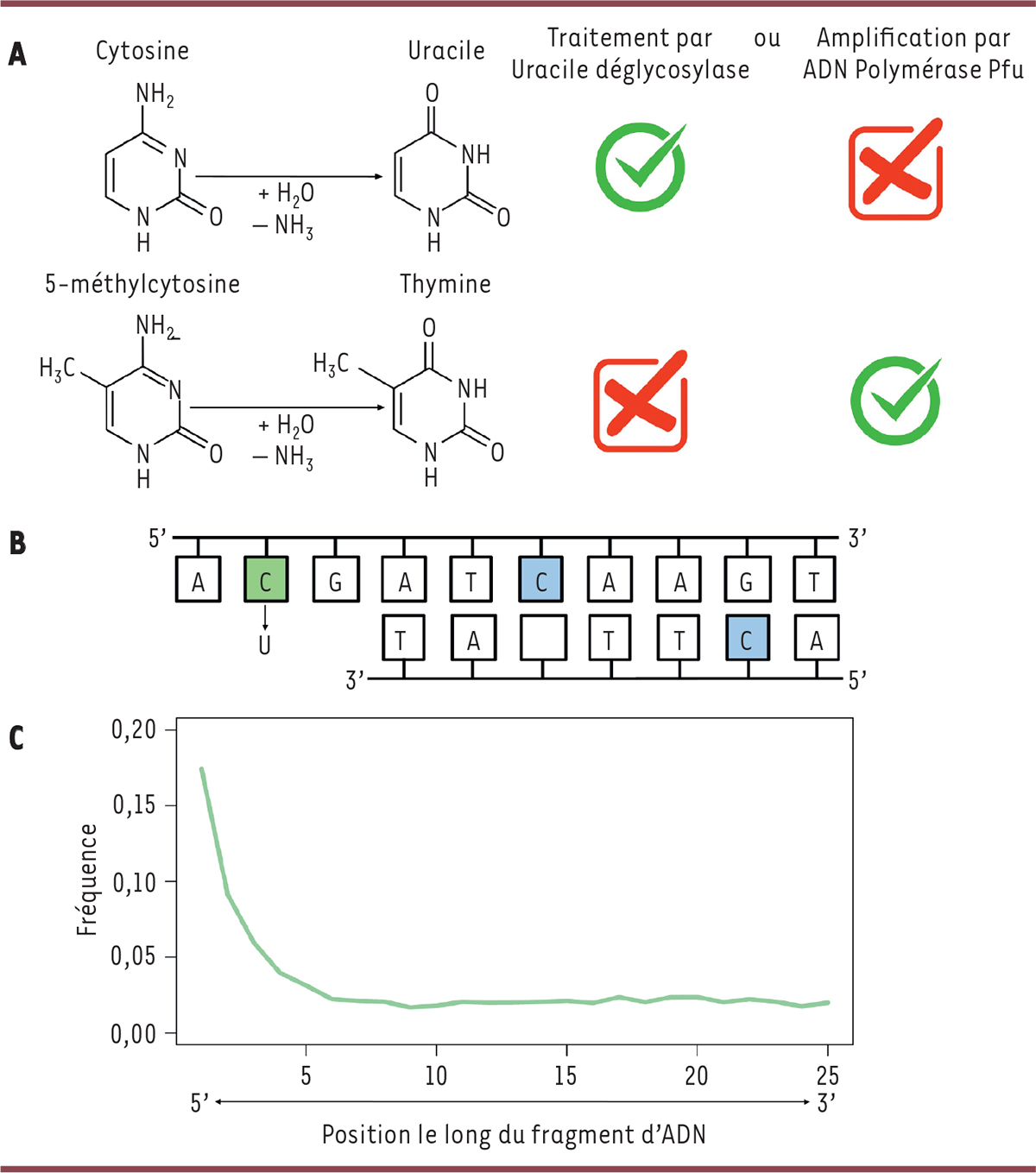
Les endommagements des bases azotées donnent accès au paléoépigénome A. La désamination des cytosines non méthylées conduit à leur transformation en uracile. Ces bases uraciles sont éliminées par un traitement avec l’uracile-ADN déglycosylase (UDG). Ces uraciles ne peuvent également pas être amplifiés par la technique de PCR (polymerase chain reaction) avec une ADN polymérase thermostable telle que l’ADN polymérase Pfu. En revanche, la désamination des cytosines méthylées conduit à leur transformation en thymines, insensibles au traitement par l’enzyme UDG, et les fragments qui les contiennent peuvent donc être amplifiés par l’ADN polymérase Pfu. B. Ces désaminations de cytosines se produisent particulièrement dans les zones où l’ADN a une structure en simple brin, à l’extrémité d’un fragment (cytosine verte), et sont beaucoup plus rares dans les zones où l’ADN a une structure en double brin (cytosines bleues). C. Cela conduit à un excès de transformations des cytosines méthylées en thymine (C→T) aux extrémités 5’ des fragments d’ADN ancien (observés ici avec le logiciel mapDamage) par rapport à une séquence d’ADN de référence.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




