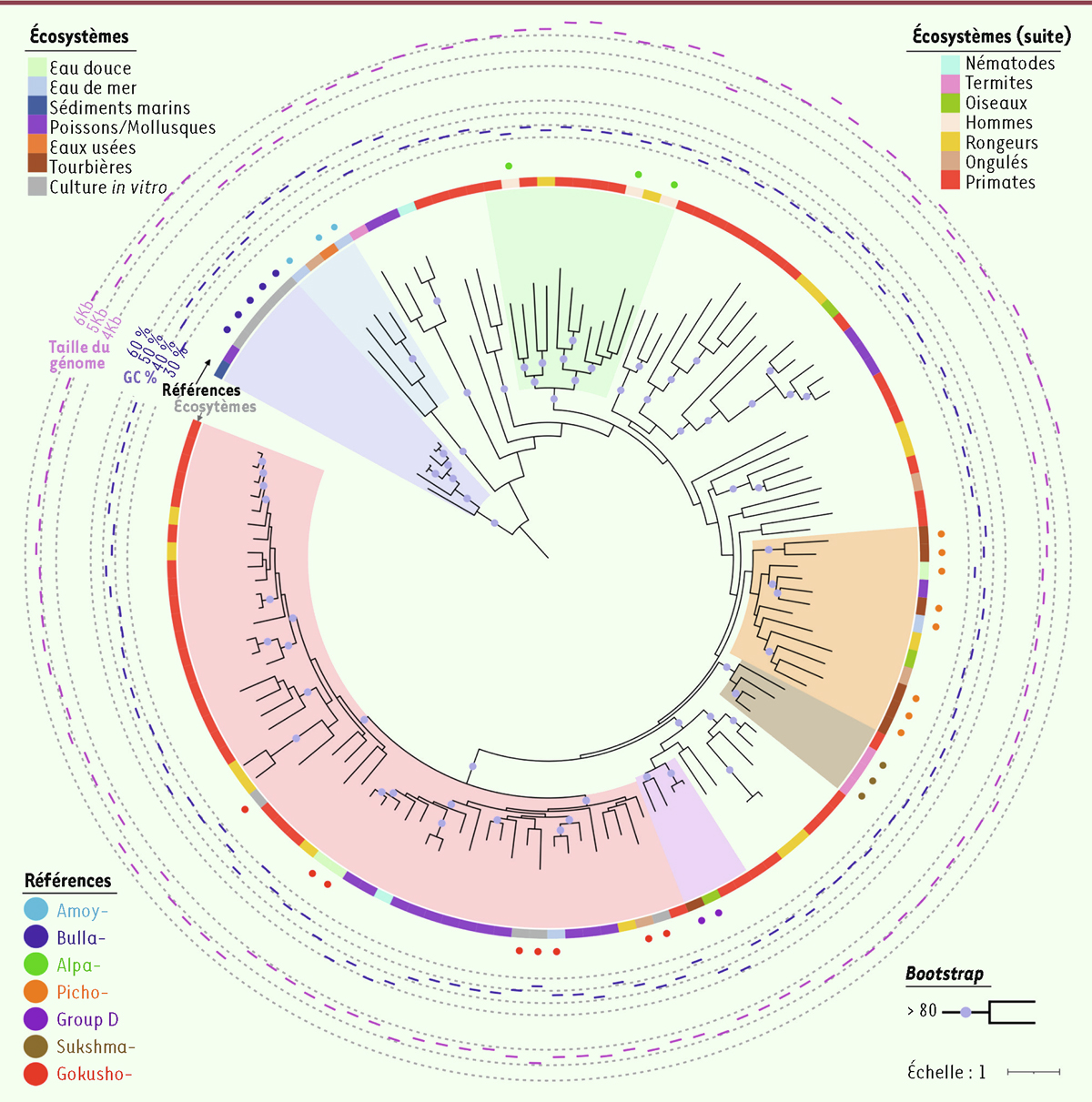
Figure 3.
Télécharger l'image originale
Phylogénie de 130 génomes de microvirus. Cette phylogénie a été réalisée à partir de la protéine de capside de 32 microvirus de références, classés précédemment dans une sous-famille connue, et 98 génomes complets assemblés à partir de 78 viromes associés à divers hôtes animaux [29]. Une valeur de « bootstrap » (pourcentage de 0 à 100 qui évalue la robustesse) est associée à chaque branche de l’arbre indiquant le nombre de fois où cette branche a été retrouvée lorsque l’on utilise différentes positions de l’alignement des protéines de capside. Les branches ayant une valeur de « bootstrap » supérieure à 80 sont indiquées avec un cercle.
À l’extérieur de la phylogénie sont présentées quatre pistes : les écosystèmes dans lesquels chaque génome a été échantillonné ; les génomes de référence, indiqués par des cercles colorés selon la sous-famille ; le pourcentage en nucléotides G et C des génomes ; et la taille de chacun des génomes. Les zones colorées indiquent les groupes monophylétiques contenant un ou plusieurs génomes de référence et éventuellement des séquences environnementales.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




