Figure 1.
Télécharger l'image originale
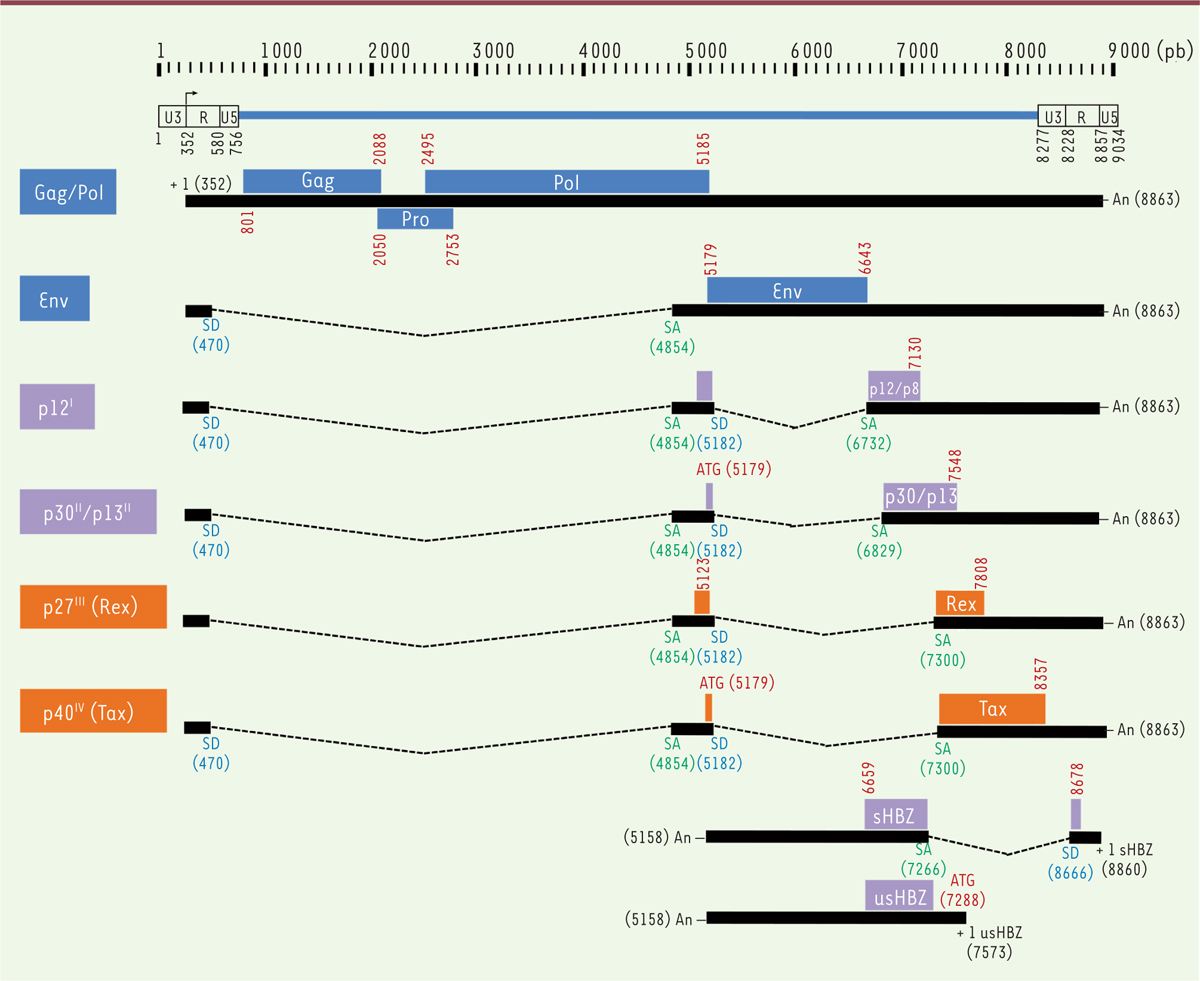
Représentation du génome intégré du HTLV-1. Le génome viral intégré dans le génome de la cellule infectée (provirus), d’une taille de 9 034 paires de bases (pb) et encadré par les deux longues répétitions terminales (ou LTR, pour long terminal repeat) est représenté en respectant l’échelle de ses différents composants. Le génome est transcrit à partir du brin sens, sous le contrôle du LTR5’ et à partir du brin antisens, sous le contrôle du LTR3’. Il code les protéines classiquement exprimées par les rétrovirus : les protéines de structure et les enzymes (Gag, Pol, Env, en bleu), mais également les protéines dites auxiliaires, dont la caractéristique est d’être nécessaires lors de l’infection in vivo mais pas pour la réplication in vitro (p30/p13, p12, p8, HBZ, en mauve), ainsi que les protéines régulatrices, dont la caractéristique est d’être nécessaires à la fois in vivo et in vitro (Rex, Tax, en orange). Les transcrits sens ou antisens sont soit non épissés (Gag/Pol, usHBZ) soit mono- (Env, sHBZ) ou bi- (p30/p13, p12/p8, Tax, Rex) épissés. Les positions des cadres ouverts de lecture et du codon d’initiation de la traduction (ATG) sont indiquées en rouge et celles des sites donneurs (SD) et accepteurs d’épissage (SA) en bleu clair et vert, respectivement. An : site de polyadénylation des transcrits.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




