Figure 1.
Télécharger l'image originale
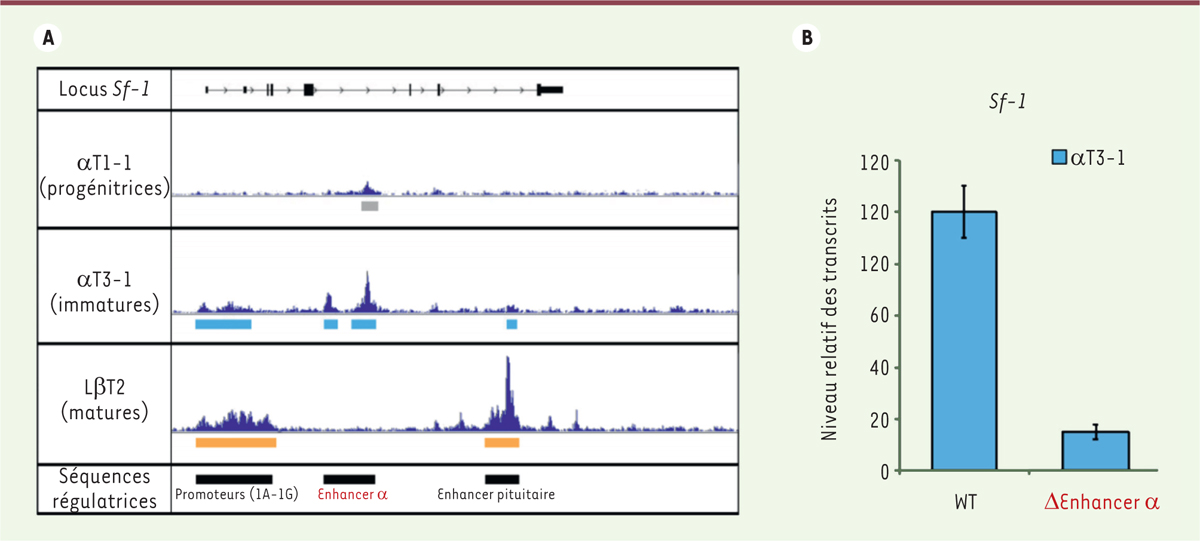
Identification de l’enhancer a, le plus précocement impliqué dans l’expression de Sf-1 au cours de la différenciation du lignage gonadotrope hypophysaire. A. « Accessibilité » de la chromatine mesurée par la technique ATAC-seq (assay for transposase accessible chromatine with high-throughput sequencing) pour le locus du gène Sf-1 dans les trois lignées modèles de cellules gonadotropes murines immortalisées : αT1-1, αT3-1 et LβT2. La hauteur des pics reflète le « degré d’ouverture » de la chromatine. Les régions accessibles de la chromatine ainsi identifiées sont soulignées sous chacune des lignées cellulaires (respectivement en gris, bleu, et jaune). La dernière ligne récapitule, en noir, l’ensemble des régions accessibles de la chromatine identifiées pour l’ensemble des lignées. B. Niveaux relatifs des transcrits de Sf-1 dans les cellules gonadotropes immatures αT3-1 témoins (wild-type, WT) ou ayant subi la délétion de l’enhancer α (DEnhancer a) mesurés par RT-PCR (reverse transcriptase-polymERαse chain reaction) après normalisation par le taux de transcrits d’un gène de ménage (Gapdh, glycERαldehyde 3-phosphate dehydrogenase). Les données correspondent à la moyenne ± SEM (standard error of the mean, ou erreur type de la moyenne) de six expériences indépendantes. Différence statistiquement significative par rapport aux cellules témoins (WT) : p < 0,001.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




