Figure 3.
Télécharger l'image originale
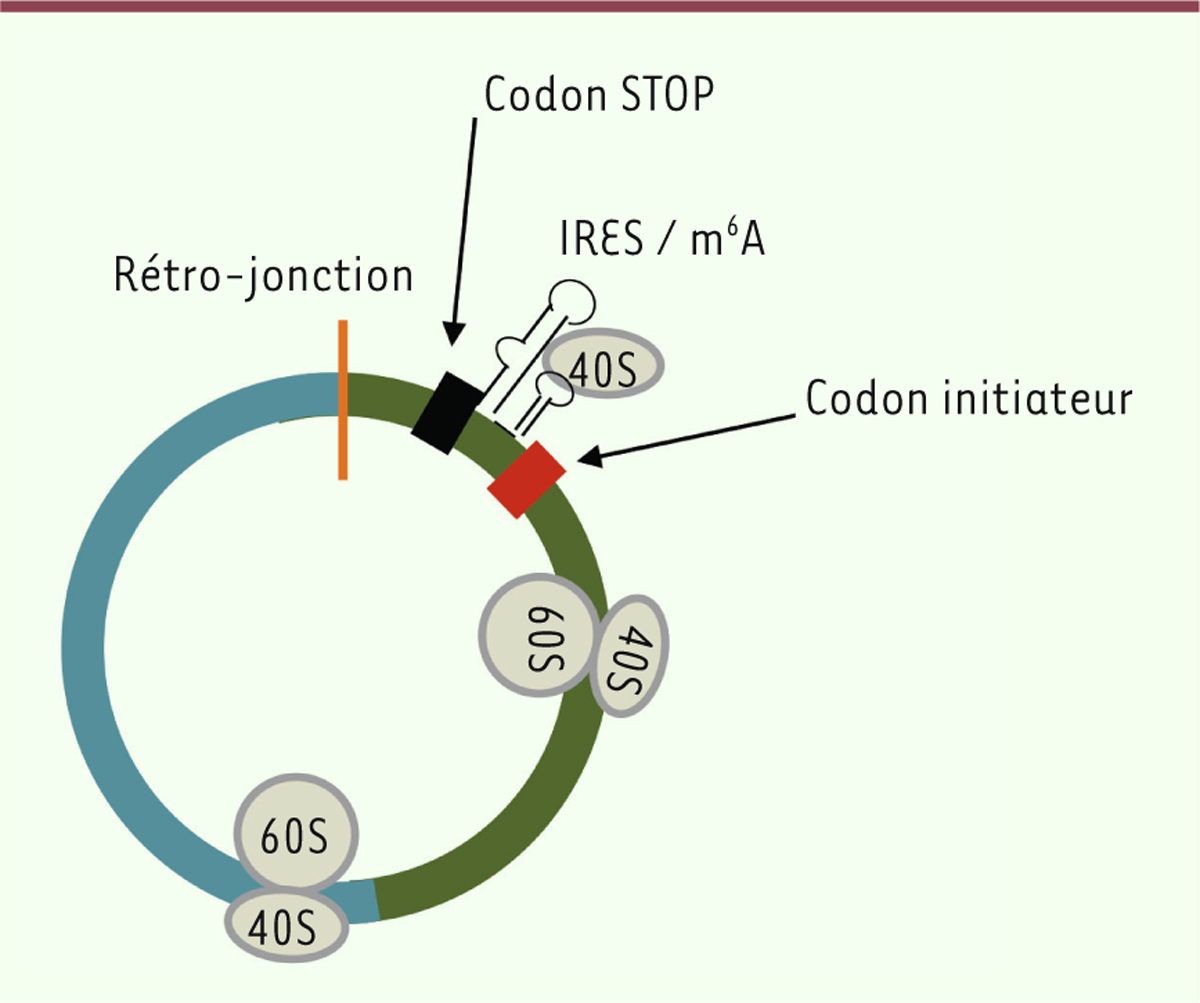
Structure caractéristique d’un circARN traduit. Dans cet exemple, l’EcARN est formé par l’assemblage de deux exons (vert, bleu). Le cadre de lecture commence au codon initiateur [boîte rouge] en direction du codon stop [boîte noire], en passant par la jonction entre les deux exons (vert > bleu), puis la rétro-jonction qui a circularisé la molécule (trait orange). Le codon stop se situe quelques nucléotides en aval de la rétro-jonction. Les séquences permettant l’initiation de la traduction (IRES/m6A) se trouvent généralement entre le codon stop et le codon initiateur. Cette configuration est celle décrite pour les circZNF609 et circMbl. La liaison interne de la sous-unité 40S du ribosome à l’EcARN, puis la traduction par le ribosome 80S après association des deux sous-unités 40S et 60S, sont schématisées. EcARN : exonic circRNA ; IRES : internal ribosome entry site ; m6A : N6-methyladenosine ; Mbl : muscleblind.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




