Figure 2.
Télécharger l'image originale
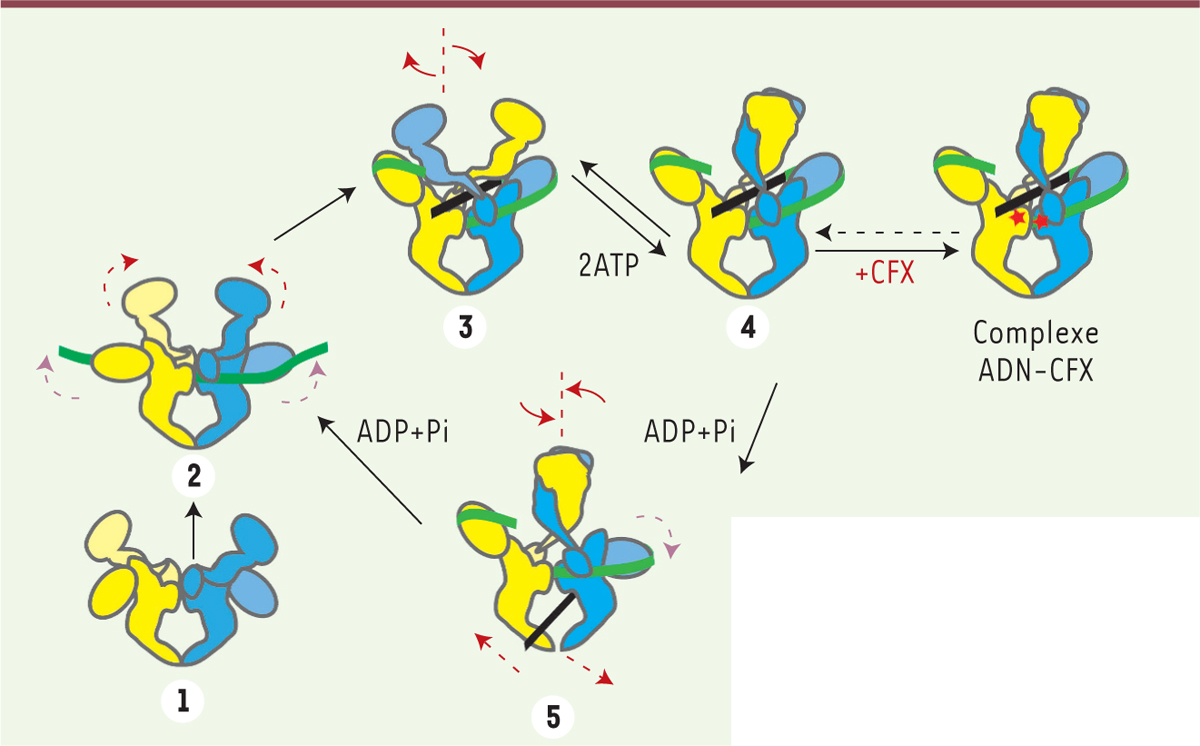
Mécanisme de surenroulement négatif par l’ADN gyrase révisé avec les nouvelles informations structurales. Les monomères de l’ADN gyrase sont colorés en jaune et bleu. (1-2) un premier segment T d’ADN se fixe à l’interface dimérique de l’ADN gyrase s’enroulant autour des domaines C-terminaux, faisant remonter les b-pinweel et l’ADN à proximité d’un des monomères du domaine ATPase. (3) Le segment T contigu au segment G est capturé à l’interface de l’enzyme et forme un croisement d’ADN stabilisé par l’entrelacement des monomères du domaine ATPase. (4) La fixation de deux molécules d’ATP entraîne la dimérisation du domaine ATPase et la fermeture du « piège à ADN » au dessus du croisement positif des segments T et G. (4-5) L’hydrolyse successive des molécules d’ATP entraîne des réarrangements structuraux qui accompagnent le transport du segment T à travers la coupure double brin du segment G. Après éjection du segment T, l’enzyme est alors prête pour un nouveau cycle catalytique. Les mouvements effectués par l’ADN gyrase peuvent être assimilés à ceux d’un nageur de crawl. L’antibiotique ciprofloxacine (CFX, étoiles rouges) s’intercale dans les coupures double brin du segment G, empêchant le passage du segment T et bloquant l’enzyme au début du cycle catalytique dans une conformation de « pré-transport » du segment T (DNA-cipro bound complex).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




