Figure 2.
Télécharger l'image originale
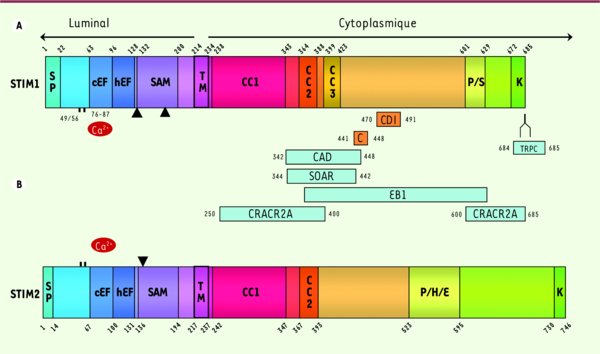
Structures des monomères STIM1 (A) et STIM2 (B). Il existe des sites de glycosylation intraluminaux en position 131 et 171 sur STIM1, et 135 sur STIM2. Des cystéines sont conservées en position 49 et 56 sur STIM1 et 53 et 60 sur STIM2. Les sites de liaison pour le Ca2+ situés à l’intérieur du RE permettant l’oligomérisation des STIM se trouvent sur le domaine cEF en position 76-87 et 80-91 respectivement pour STIM1 et STIM2. En (A) : sites de liaison sur STIM1 des protéines de la signalisation calcique : CDI, domaine d’inactivation dépendante du Ca2+ de Icrac contenant un cluster de 7 acides aminés (aa) entre les positions 475 et 483 modulant positivement et négativement le degré d’inactivation. S/P peptide signal ; cEF et hEF, motifs permettant la liaison du Ca2+ ; SAM, sterile alpha-motif ; TM, segment transmembranaire ; CC1, CC2 et CC3, domaines coiled-coil ; P/S, domaine riche en proline et sérine et P/H/E, riche en proline et histidine ; K domaine riche en acides aminés polybasiques. C, canaux calciques dépendants du potentiel Cav1.2 ; CAD, domaine d’activation du Icrac ; SOAR (STIM-ORAI activation region), région de couplage STIM1-ORAI1 ; EB1 (microtubule plus end tracking protein) ; CRACR2A (CRAC regulator 2A) ; canaux TRPC (transient receptor potential canonical) 1, 3, 4, 5 et 6. Les SERCA se disposent autour de STIM1 après vidange calcique du RE, mais il n’y a pas d’interactions visibles entre ces deux protéines.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




