Figure 2
Télécharger l'image originale
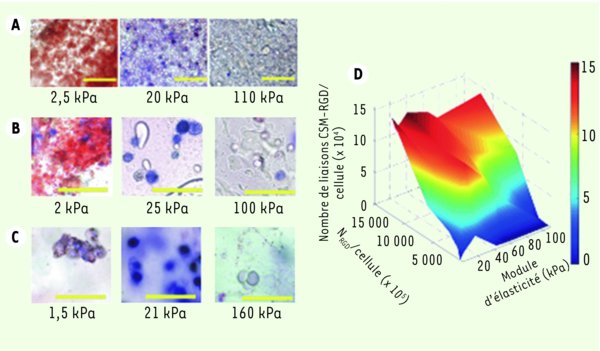
La configuration de la matrice 3D altère le devenir des CSM. A-C. Coloration in situ montrant l’expression de l’activité de la phosphase alcaline (PAL) (Fast Blue, biomarqueur ostéogénique de couleur bleue) et l’accumulation de lipides neutres (Oil Red O, biomarqueur adipogénique de couleur rouge) après 1 semaine en culture en présence de facteurs ostéogéniques et adipogéniques CSM encapsulées dans différentes matrices polymères. Ces dernières étaient modifiées par des greffes de la séquence peptidique RGD (754 μM) en concentration constante, et présentant différentes rigidités (différentes valeurs de kPa) : alginate-RGD (A), agarose-RGD (B) ou poly (éthylène glycol diméthacrylate)-RGD (C). D. Vue en 3D de Nb (mesure du nombre de liaisons CSM-RGD par cellule obtenue à partir de la technique FRET) en fonction du module d’élasticité (E) et de NRGD/cellule (qui reflète la densité en RGD). Les résultats indiquent qu’un nombre important de cellules de la population naïve des CSM encapsulées dans des matrices de rigidité avoisinant 11-30 kPa produisent, indépendamment de la nature de la matrice polymère, de la phosphatase alcaline (PAL) et de l’ostéocalcine (OCN), deux marqueurs indicatifs de l’ostéogenèse. De plus, la relation biphasique entre Nb et la rigidité a été maintenue lorsque E a été modifiée, suggérant que le nombre de points d’attachement RGD-cellules est aussi un paramètre important corrélé à la spécification ostéogénique (reproduit de la ref [7] avec la permission des auteurs).
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




