Figure 2.
Télécharger l'image originale
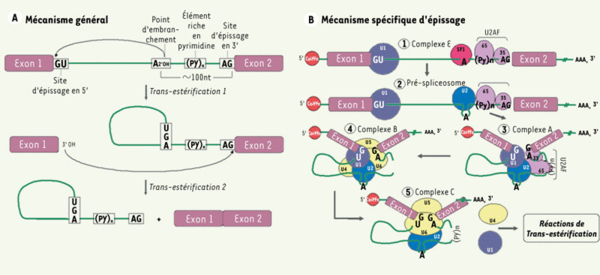
Épissage. A. Mécanisme général. 1. L’épissage est dirigé par des séquences consensus qui sont situées à la jonction exon/intron (site d’épissage en 5’) et à la jonction intron/exon (site d’épissage en 3’). Au site d’épissage en 5’, on trouve la séquence AG½GURAGU (barre verticale : site d’épissage; nucléotides soulignés : séquence invariable; R : purine). Un point d’embranchement constitué d’une séquence dont le résidu adénosine est hautement conservé : YNCURAY (Y : pyrimidine ; N : nucléotide aléatoire) et qui est suivi d’un élément riche en pyrimidine est présent environ 100 nucléotides avant le site d’épissage en 3’. Le site d’épissage en 3’ de l’intron est caractérisé par le motif YAG½RNNN.2. Le clivage de séquences non codantes est un processus nucléaire réalisé par deux étapes de trans-estérifications. Ainsi, la liaison 3’-5’ phosphodiester du résidu guanosine présent au site d’épissage en 5’ subit une attaque nucléophile par le résidu adénosine hautement conservé trouvé au point d’embranchement. Il y a alors formation d’une interaction covalente entre ces deux nucléotides permettant à l’intron d’adopter la forme d’un lasso. L’extrémité 3’ libre de l’exon en amont de l’intron à épisser réalise la deuxième trans-estérification par l’attaque nucléophile de la liaison 3’-5’ phosphodiester du résidu guanosine au site d’épissage en 3’. Il y a alors détachement de l’intron déjà sous forme de lasso et liaison entre les deux exons adjacents.B. Mécanisme spécifique. Les deux étapes de trans-estérification sont rendues possibles grâce à un complexe catalytique macromoléculaire nommé spliceosome. Le spliceosome est constitué de cinq petites ribonucléoprotéines nucléaires snRNP (small nuclear ribonucleoprotein). Chacune d’elles est constituée d’un petit ARN nucléaire associé à plusieurs protéines pour former les snRNP U1, U2, U4, U5 et U6. De nombreuses protéines accessoires sont également présentes dans le spliceosome dont les protéines SF1 et le dimère U2AF. La formation du spliceosome sous forme active doit préalablement se réaliser afin de permettre l’excision intronique via les deux étapes de trans-estérification. Pour ce faire, un assemblage en cinq étapes est nécessaire. Il débute par la formation du complexe E où il y a appariement de U1 au site d’épissage en 5’, de SF1 au résidu adénosine très conservé à hauteur du point d’embranchement, de U2AF-65 à l’élément riche en pyrimidine et de U2AF-35 au site d’épissage en 3’. Par la suite, le pré-spliceosome est formé par la délocalisation de SF1 après la liaison de U2 au point d’embranchement. Cette association n’inclut toutefois pas le résidu adénosine afin qu’il soit disponible pour la première attaque nucléophile. Le complexe A apparaît grâce à la réorganisation du pré-spliceosome qui implique la formation d’interactions entre les facteurs U1, U2 et U2AF. Cela contribue au rapprochement des exons favorisant leur éventuelle liaison. L’arrivée du tri-snRNP U4/U5/U6 au sein des séquences consensus d’épissage entraîne la formation du quatrième complexe, le complexe B. Sa présence permet de libérer le point d’embranchement de même que le site d’épissage en 3’. Finalement, le complexe catalytique, ou complexe C, est formé par l’association de U6 et U2 au site d’épissage en 5’ déclenchant le départ de U1 et U4. Les étapes de trans-estérification peuvent alors avoir lieu.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




