Figure 1.
Télécharger l'image originale
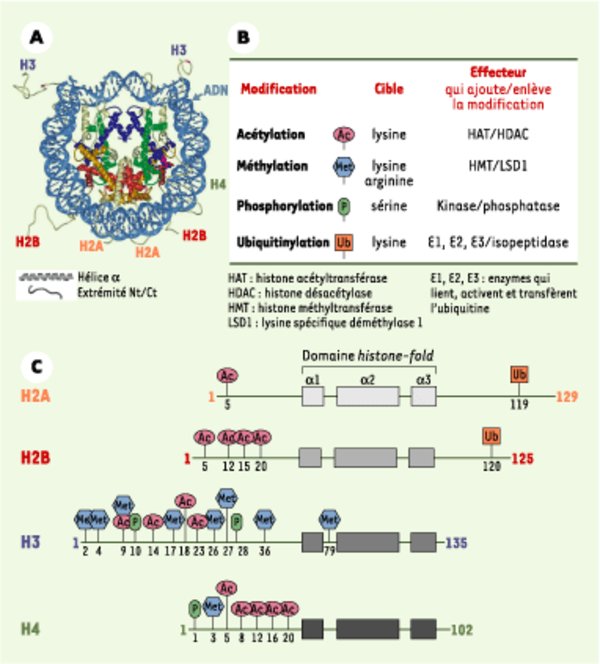
Particule cœur du nucléosome et modifications des histones. A. Structure de la particule cœur du nucléosome. La double hélice d’ADN (en bleu clair) s’enroule autour d’un octamère protéique constitué de deux molécules de chaque histone : H2A (en jaune), H2B (en rouge), H3 (en bleu) et H4 (en vert). Nt/Ct indique les extrémités amino- et carboxyterminales (d’après [3]). B. Les extrémités amino- et carboxyterminales des histones sont les cibles privilégiées de modifications post-traductionnelles : les mieux caractérisées sont l’acétylation, la méthylation, la phosphorylation et l’ubiquitinylation. Leurs cibles et leurs effecteurs (enzymes qui catalysent l’ajout ou la suppression de la modification) sont indiqués. Une seule histone déméthylase a été décrite à ce jour, LSD1 (lysine specific demethylase 1), qui est capable de déméthyler la lysine 4 de l’histone H3. C. Structure et modifications des histones majeures de la particule cœur du nucléosome chez l’homme. Les trois hélices α du motif conservé histone-fold, intervenant dans la dimérisation des histones, sont représentées par des rectangles gris. La nature et la position des modifications post-traductionnelles sont schématisées comme en B.
Current usage metrics show cumulative count of Article Views (full-text article views including HTML views, PDF and ePub downloads, according to the available data) and Abstracts Views on Vision4Press platform.
Data correspond to usage on the plateform after 2015. The current usage metrics is available 48-96 hours after online publication and is updated daily on week days.
Initial download of the metrics may take a while.




