Figure 1.
Télécharger l'image originale
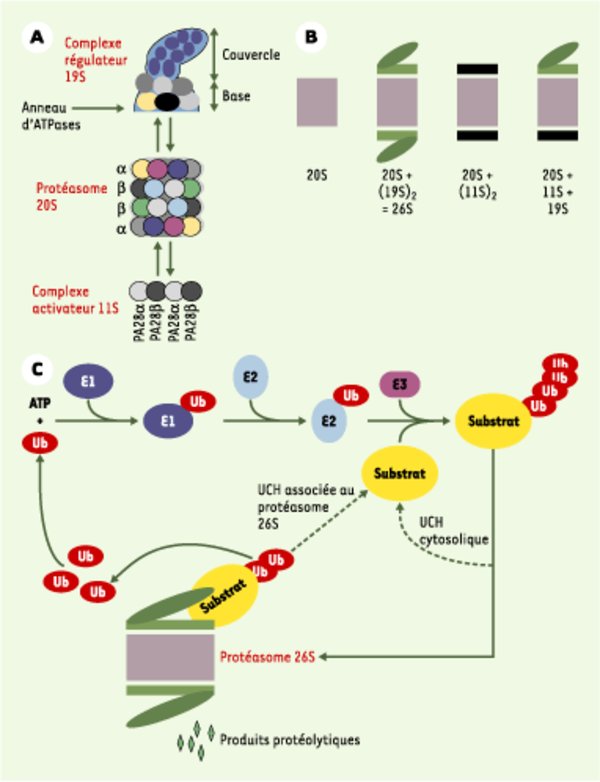
Protéasomes et voie ubiquitine. A. Le protéasome 20S et ses complexes régulateurs. Le protéasome 20S est le cœur catalytique de la machinerie protéasomique. Il s’agit d’un cylindre creux formé de 4 anneaux : 2 anneaux identiques de 7 sous-unités de type α et 2 autres, également identiques entre eux, de 7 sous-unités de type β. Les activités protéolytiques sont situées à l’intérieur de la structure et sont portées par 3 des 7 sous-unités β. Les anneaux α assurent l’interaction avec les complexes régulateurs du protéasome 20S et permettent l’entrée des substrats dans la chambre catalytique grâce à leur orifice central. Le complexe activateur 19S participe à la dégradation des protéines ubiquitinylées grâce à sa capacité à reconnaître les chaînes d’ubiquitine. Il possède un anneau d’ATPases chaperons participant à la déstructuration des substrats et, peut-être, à leur injection dans la cavité protéolytique, ainsi qu’une activité de désubiquitinylation (activité UCH, ubiquitin C-terminal hydrolase). Le complexe régulateur 11S est un anneau de 6 à 7 protéines de deux types différents : PA28α (proteasome activator) et PA28β. Il modifie les propriétés catalytiques du 20S, mais n’est pas capable de reconnaître des protéines ubiquitinylées. B. Les différents types de protéasome. Le protéasome 20S, retrouvé en partie libre dans la cellule, peut également s’associer avec des complexes 19S et 11S à chacune de ses extrémités. La particule traditionnellement appelée protéasome 26S est composé d’un 20S et de deux 19S. Les différentes particules sont présentes en quantités comparables dans les cellules mammifères. C. La voie ubiquitine/protéasome. L’ubiquitinylation des protéines fait intervenir trois types d’enzyme. E1 (ubiquitin-activating enzyme) est unique. Elle active l’ubiquitine et la transfère sur des enzymes E2 (ubiquitin-conjugating enzymes), qui sont au nombre d’une trentaine chez les mammifères. Directement, ou avec l’aide de composants E3, elles transfèrent et polymérisent l’ubiquitine sur les substrats. Dans la majorité des cas, les facteurs E3, dont certains sont multimériques, forment un pont moléculaire entre E2 et le substrat. D’autres transfèrent eux-mêmes l’ubiquitine sur les protéines acceptrices. Les E3 assurent l’essentiel de la spécificité de la réaction grâce à leur grand nombre (plusieurs centaines de E3 différentes dans une cellule eucaryote), bien qu’il existe une certaine redondance dans le système. Ainsi, une même protéine peut être reconnue par différentes E3 et une même E3 peut reconnaître différents substrats. L’ubiquitinylation est réglée par la signalisation intracellulaire via des modifications post-traductionnelles des substrats (phosphorylations, hydroxylation…) ou au travers de la modulation de l’activité des E3. Les protéines ubiquitinylées peuvent être désubiquitinylées par des UCH cytosoliques et recyclées. Dans le cas contraire, elles sont reconnues par des protéasomes contenant un complexe régulateur 19S qui assure la désubiquitinylation, la déstructuration et l’acheminement vers la cavité protéolytique. Si elles sont insuffisamment ou improprement ubiquitinylées, les protéines peuvent être recyclées grâce à une activité UCH portée par le complexe 19S lui-même. L’ubiquitine, qui est très stable, est réutilisée.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




