Figure 4.
Télécharger l'image originale
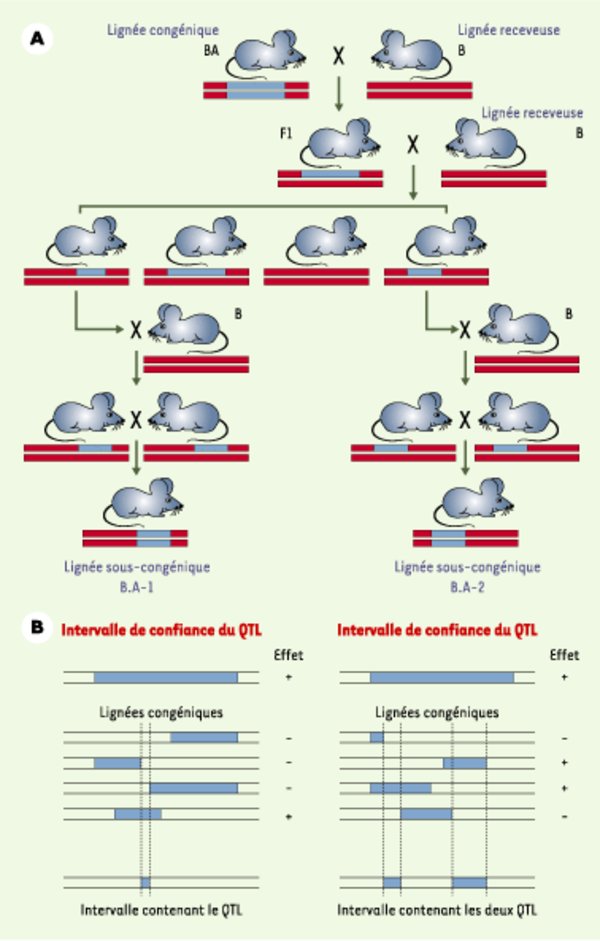
Utilisation de lignées sous-congéniques pour affiner la localisation d’un QTL (quantitative trait locus). A. À partir d’une lignée congénique porteuse d’un grand segment chromosomique, on dérive plusieurs lignées sous-congéniques portant des segments partiellement chevauchants : la lignée congénique (BA) est croisée deux fois avec la lignée receveuse (B) pour produire une population de quelques dizaines d’individus qui sont génotypés pour des marqueurs couvrant le segment initial. Les événements de recombinaison chez le parent F1 ont produit des chromosomes contenant un segment congénique plus court. Les individus qui les portent sont accouplés avec la lignée receveuse et leurs descendants hétérozygotes pour le même segment sont croisés entre eux pour obtenir une lignée sous-congénique homozygote. B. L’étude de lignées sous-congéniques permet de confirmer les résultats d’une analyse QTL effectuée sur un croisement, en comparant la composition génétique de chaque lignée avec ses propriétés pour le caractère étudié (« effet »). À gauche : les résultats permettent de confirmer l’existence d’un QTL et de réduire l’intervalle dans lequel il doit être recherché. À droite : l’analyse des lignées montre que l’intervalle défini initialement contient en réalité deux QTL liés, combinant leurs effets (aucun sous-intervalle ne permettant à lui seul d’expliquer l’ensemble des résultats, l’effet global est dû à l’effet de l’un et/ou de l’autre QTL), qui ont pu être isolés dans deux lignées sous-congéniques.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




