Figure 1.
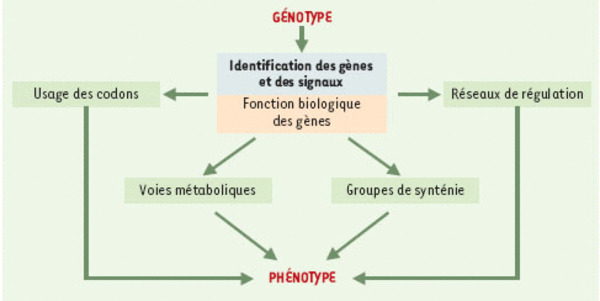
Stratégies d’annotation in silico des génomes. L’annotation des séquences génomiques consiste à extraire, à partir d’outils informatiques, le maximum d’information des données de séquences afin de prédire des caractéristiques phénotypiques permettant de guider le travail expérimental. Dans une première étape, les régions codantes et les signaux (promoteurs et terminateurs de la transcription, ribosome binding site ou signal RBS) sont recherchés. Les gènes identifiés sont alors traduits en séquences peptidiques et la mise en œuvre de méthodes fondées sur la recherche de similarités avec les protéines répertoriées dans les banques de séquences permet de caractériser la fonction biologique de près de la moitié d’entre eux. On s’intéresse alors, dans un deuxième temps, aux relations qui lient ces objets biologiques : caractérisation des réseaux de régulation, des groupes de synténie, des voies métaboliques, et de l’usage des codons des gènes des organismes étudiés.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.




